Des séquences d'acides nucléiques identifiées en une seule étape
Des chercheurs du LAAS-CNRS ont réalisé un capteur nanofluidique capable de discriminer, en une seule étape, des séquences de nucléotides (ARN, ADN) qui ne diffèrent que par une seule paire de bases. La technologie pourrait être appliquée au dosage de biomarqueurs moléculaires de certains cancers. Ces résultats sont publiés dans la revue ACS Sensors.
Le dosage de biomarqueurs moléculaires dans le sang pour améliorer la prise en charge de certains cancers est un domaine de recherche en plein développement. Une équipe du Laboratoire d’analyse et d’architecture des systèmes (LAAS-CNRS), en collaboration avec des chercheurs du Centre de recherches en cancérologie de Toulouse (CRCT, Inserm/CNRS), a mis au point une puce nanofluidique qui permet d'identifier rapidement, et en une seule étape, une séquence d'ARN ou d'ADN courts, avec une précision d'une seule paire de bases.
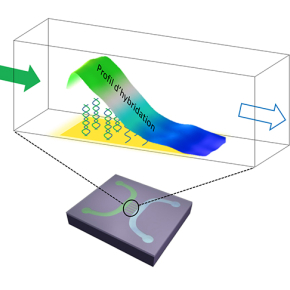
La particularité de ce capteur fluidique est de comporter, dans sa partie centrale, un canal de dimension nanométrique : plusieurs centaines de micromètres de longueur, mais seulement 500 nanomètres de hauteur. Dans ce canal ont été fixées des molécules sondes, capables d'interagir spécifiquement avec les molécules cibles que l'on veut détecter. La puce a été réalisée en salles blanches, grâce aux techniques et au savoir-faire du LAAS-CNRS en matière de microfabrication. La dimension nanométrique du capteur est un point clé, car c'est ce milieu confiné qui favorise une vitesse de réaction non uniforme le long du nanocanal. L'observation des interactions molécules sondes/molécules cibles, à l'aide d'un microscope à fluorescence, donne un profil spatial qui est la signature de la molécule que l'on veut détecter. Le capteur nanofluidique a le grand avantage d'identifier une molécule cible en temps réel et en une seule étape d’injection de solution.
À titre de démonstration, les chercheurs ont pu ainsi discriminer deux microARNs, petites molécules d’ARN non codant comprenant une vingtaine de paires de bases pouvant faire office de marqueurs du cancer, et qui ne diffèrent que par une seule paire de bases. La puce nanofluidique, qui a été développé dans le cadre du projet Oncodevice*, ouvre donc la voie à la détection fine de biomarqueurs, en particulier de segments d'ADN ou d'ARN circulant dans le sang. Cette collaboration se poursuit entre le Centre de recherches en cancérologie de Toulouse, qui travaille notamment à la mise au point de nouveaux biomarqueurs moléculaires non invasifs du cancer du pancréas, et l'équipe du LAAS-CNRS, qui cherche désormais à s'affranchir du marquage fluorescent des molécules cibles, et à améliorer la sensibilité du dispositif, avec comme objectif commun d’assurer un transfert de technologie plus rapide vers la clinique.
*Ces travaux ont été réalisés dans les salles blanches du réseau Renatech (https://www.renatech.org/ ).
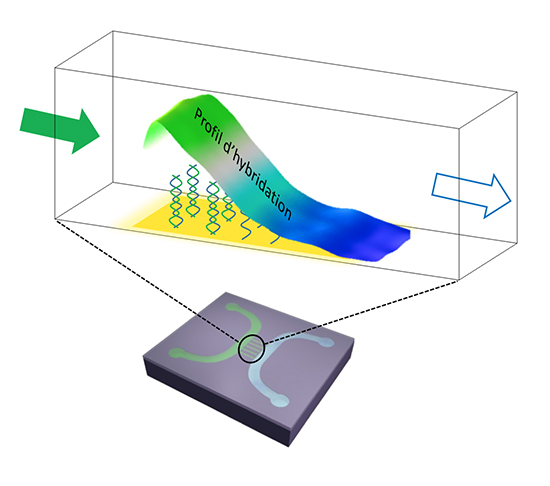
Principe de fonctionnement du biocapteur nanofluidique : la réduction des dimensions du canal fluidique entraine une hybridation non uniforme entre les molécules cibles injectées et les molécules immobilisées dans le canal le long du capteur. L’analyse du profil d’hybridation au cours du temps permet d’identifier rapidement les molécules cibles.
Références :
Spatial Analysis of Nanofluidic-Embedded Biosensors for Wash-Free Single-Nucleotide Difference Discrimination
J. Cacheux, M. Brut, A. Bancaud, P. Cordelier, T. Leïchlé,
ACS Sensors, 2018, 3 (3), pp 606–611
DOI: 10.1021/acssensors.7b00667