Une nouvelle interaction découverte dans la régulation du fonctionnement des cellules
De nombreuses machineries moléculaires, impliquant des protéines spécifiques, gèrent l’état cellulaire. Alors que leurs rôles pourraient sembler très compartimentés, des scientifiques du laboratoire IMoPA, de l’IPHC, du laboratoire Génétique des génomes et de l’IGH ont découvert un nouveau cas d’interaction génétique et physique entre des machineries aux fonctions distinctes. Publiés dans la revue Nature Communications, ces travaux multidisciplinaires montrent l’étendue des régulations contrôlant le fonctionnement cellulaire.
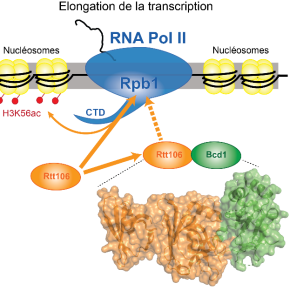
Alors que la recherche fondamentale explore avec de plus en plus d’acuité la myriade d’interactions entre les gènes ou les protéines qui en sont issues, des liens inattendus émergent entre des mécanismes biologiques jusqu’alors étudiés séparément. Les snoRNP sont ainsi de petits complexes moléculaires d’ARN et de protéines qui participent à la formation des ribosomes, les machines cellulaires chargées de décoder l’ARN messager afin de synthétiser des protéines. De son côté, la chromatine est la structure qui compacte l’ADN au sein des cellules. Elle constitue en cela une grande partie des chromosomes et est formée de deux types de protéines : les histones et les non-histones. La structure de la chromatine joue un rôle majeur dans l’expression des gènes : elle est régulée par de nombreux acteurs protéiques qui contrôlent l’accessibilité de l’ADN, et donc la possibilité pour la cellule d’exprimer ou non ses gènes. Des chercheurs et chercheuses du laboratoire Ingénierie moléculaire et physiopathologie articulaire (IMoPA, CNRS/Université de Lorraine), de l’Institut pluridisciplinaire Hubert Curien (IPHC, CNRS/Université de Strasbourg), du laboratoire Génétique des génomes (CNRS/Institut Pasteur) et de l’Institut de génétique humaine (IGH, CNRS/Université de Montpellier) ont découvert l’existence de liens génétiques, physiques et fonctionnels entre des protéines permettant l’assemblage des snoRNP, dont la protéine Bcd1, et des gènes codant une protéine (Rtt106) qui modèle la chromatine.
Afin de mener ces travaux, les scientifiques ont choisi comme modèle la levure Saccharomyces cerevisiae. Comme ce modèle biologique présente des facilités de culture en laboratoire, le criblage génétique permettant d’identifier des gènes a pu être automatisé à haut débit. Après une partie de criblage in vivo, une phase in vitro a aidé à décrire à l’échelon atomique les éléments clés de l’interaction entre les protéines Bcd1 et Rtt106. Fort de ses résultats, de nouvelles hypothèses ont ensuite été testées in vivo, révélant l’existence d’un mécanisme de compétition. L’équipe a ainsi proposé un modèle où l’activité d’une des protéines impliquées dans le remodelage de la chromatine est contrôlée par une protéine chargée de l’assemblage de certaines snoRNP. De tels liens, entre des processus biologiques aussi distincts, sont rarement mis en évidence. Ces travaux renforcent ainsi l’émergence de tout un pan de recherche, qui pourrait aboutir à la découverte de mécanismes de régulation génétique encore inconnus.
© Bragantini et al.
Références
The box C/D snoRNP assembly factor Bcd1 interacts with the histone chaperone Rtt106 and controls its transcription dependent activity.
B. Bragantini, C. Charron, M. Bourguet, A. Paul, D. Tiotiu, B., Rothé, H. Marty, G. Terral, S. Hessmann, L. Decourty, ME. Chagot, JM. Strub, S. Massenet, E. Bertrand, M. Quinternet, C. Saveanu, S. Cianférani, S. Labialle, X. Manival, B. Charpentier.
Nature Communications 2021 March 25.
https://doi.org/10.1038/s41467-021-22077-4