Des enzymes caractérisées in vivo grâce à la biologie synthétique et à la biologie des systèmes
Une équipe de scientifiques toulousains a développé une stratégie pour mesurer in vivo des paramètres enzymatiques dans leur contexte cellulaire. Cette méthode innovante, qui utilise l'ingénierie génétique et les outils de modélisation et d'analyse de la biologie des systèmes, pourrait être utilisée pour optimiser des procédés de production biotechnologiques. Les résultats de l'étude sont publiés dans The Embo Journal.
Les enzymes sont des protéines qui catalysent presque toutes les réactions chimiques dans les organismes vivants, et jouent un rôle crucial dans le fonctionnement des cellules. Mais l'étude de ces catalyseurs - l'enzymologie - est le plus souvent réalisée in vitro, dans des conditions diluées et contrôlées. L'efficacité des enzymes est ainsi évaluée dans des conditions très éloignées du milieu cellulaire réel, complexe et hétérogène, dans lequel la diffusion des molécules peut varier, et qui peut produire des effets de compartimentation (séparation physique des molécules mises en jeu).
Pour mieux comprendre les mécanismes enzymatiques en jeu dans les cellules, et évaluer leur efficacité dans des conditions réelles, des scientifiques de Toulouse Biotechnology Institute (TBI, CNRS/INRAE/INSA Toulouse) ont mis au point une méthode pour mesurer in vivo les paramètres enzymatiques dans leur contexte cellulaire. L'étude a été réalisée en prenant comme modèle la production de caroténoïdes chez la levure Saccharomyces cerevisiae, un micro-organisme fréquemment utilisé en biotechnologies, et s'est focalisée sur le rôle d'enzymes membranaires difficiles à étudier in vitro : la phytoène synthase (CrtB) et la phytoène désaturase (CrtI).
Deux techniques de pointe ont été mobilisées pour ce projet. Avec les outils d'ingénierie génétique de la biologie synthétique, les scientifiques ont créé une large gamme de souches de levures modifiées, chacune produisant une concentration différente de la molécule de départ (le ''substrat'' de la réaction catalytique). Des mesures intracellulaires de la formation des produits de la réaction et des concentrations en enzymes ont permis de calculer les paramètres cinétiques caractérisant les enzymes étudiées. Par ailleurs, un modèle mathématique du fonctionnement de l'enzyme, basé sur la biologie des systèmes (qui prend en compte toutes les molécules intervenant dans le processus), a servi à analyser et interpréter les données. Résultat : les paramètres enzymatiques mesurés in vivo peuvent diverger de ceux obtenus in vitro.
Cette méthode, fiable et polyvalente, devrait permettre de mesurer les paramètres cinétiques des enzymes dans différents environnements cellulaires. C'est donc un nouvel instrument pour mieux comprendre les mécanismes enzymatiques dans les cellules biologiques. C'est aussi un moyen innovant pour régler de manière optimale les concentrations de la molécule de départ et de l'enzyme qui catalyse la réaction chimique, ce qui devrait intéresser les spécialistes des procédés de productions biotechnologiques.
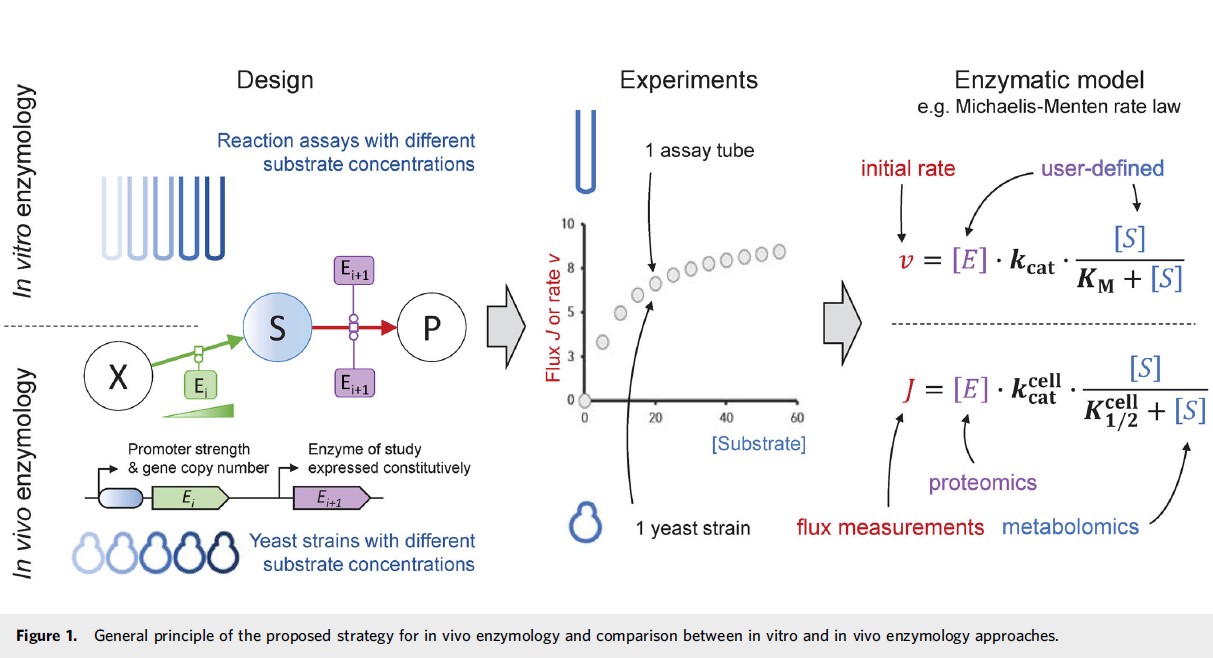
In vitro (en haut) : les paramètres d'une réaction enzymatique sont déterminés en mesurant la vitesse (v) de la réaction à différentes concentrations de substrat (S, molécule de départ de la réaction). Chaque tube réactionnel contient une concentration de substrat et pour chacune, la vitesse de la réaction est mesurée. La courbe permet d'obtenir trois paramètres importants : KM, Vmax and kcat.
In vivo (en bas) : l'ingénierie génétique permet de créer des souches de levures avec différentes quantités de l'enzyme (Ei) qui produit le substrat de la réaction. Chaque souche construite correspond à une concentration de substrat. Pour chacune d’entre elles, on peut mesurer le flux (J) de la réaction catalysée et calculer les équivalents in vivo des paramètres enzymatiques : Kcell1/2, Vcellmax et kcellcat.
© CC0 1.0
Références
Combining systems and synthetic biology for in vivo enzymology.
Sara Castaño-Cerezo, Alexandre Chamas, Hanna Kulyk, Christian Treitz, Floriant Bellvert, Andreas Tholey, Virginie Galéote, Carole Camarasa, Stéphanie Heux, Luis F Garcia-Alles, Pierre Millard & Gilles Truan.
EMBO Journal, publié le 25 septembre 2024.
https://doi.org/10.1038/s44318-024-00251-w
Article disponible sur la base d’archives ouvertes HAL